以前、VCFファイルから必要なデータを抽出する方法として、Rを使う方法を少し書きました。複雑な操作をする時はRが便利と思いますが、比較的簡単な操作(たとえば特定の情報を持つSNPsの行を抽出する場合など)を行う場合は、UNIXのコマンドである"grep"を利用することで比較的簡単にデータ抽出を行うことができます。grepは、指定したファイル内を検索して、マッチする文字列を含む行を返すコマンドです(詳しくはこちらなど参照)。
ここでは、grepを用いたデータ抽出の方法を紹介します。
解析を行う前に、Mac OS Xの場合は、より高速に動作するGNU grepをインストールします(OS XにはBSD grepがすでに入っていますが、こちらよりGNU grepのほうが速いらしい)。
Homebrewを使用します。
brew update #brewをupdateする brew tap homebrew/dupes brew install homebrew/dupes/grep
無事インストールされているかどうかを確認するため、以下のコマンドを入力します。
which ggrep
うまくインストールされていると"/usr/local/bin/ggrep"など、ggrepのPATHが表示されます。
また、versionの確認は以下のコマンドで。
ggrep --version
GNU grepのダウンロードに際して、参考にしたページは以下です。
(2) サンプルVCF fileのダウンロード
ここでは、Variant Effect PredictorなどでアノテーションされたVCFファイルのサンプルとして以下のサイトから"MHC_test4.var.annotated.vcf"というファイルをダウンロードして下さい。次に、適当な場所にフォルダを作成し、その中にサンプルVCFファイル(MHC_test4.var.annotated.vcf)を保存して、そのフォルダに移動して下さい。
(3) grepによるデータの抽出その1: 特定の遺伝子上にあるSNPs
例えば、HLA-AのSNPsを検索する場合、ggrepで以下のように実行します。
ggrep -w HLA-A MHC_test4.var.annotated.vcf > HLA-A.vcf
結果のファイルHLA-A.vcfの中身を見ると、以下のようになっています。
less HLA-A.vcf # HLA-A.vcfの中身を表示 6|29941244|.|C|G|999|.|DP=138;VDB=0.0000;AF1=0.3334;AC1=2;DP4=58,11,48,20;MQ=49;FQ=403;PV4=0.068,0.36,0.37,0.38;CSQ=G|upstream_gene_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||||||||||rs2571420|16|1|HGNC|HGNC:4931||||G:0.0790|G:0.08|G:0.05|G:0.02||G:0.14|||||||||||GT:PL:GQ|1/1:255,205,0:99|0/0:0,33,191:33|0/0:0,175,255:99 6|29941256|.|A|G|31.9|.|DP=150;VDB=0.0001;AF1=0.3229;AC1=2;DP4=101,36,9,1;MQ=50;FQ=33.5;PV4=0.45,4.9e-34,1,1;CSQ=G|upstream_gene_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||||||||||rs9260078|4|1|HGNC|HGNC:4931||||A:0.2897|G:0.79|G:0.76|G:0.74||G:0.62|||||||||||GT:PL:GQ|0/0:0,220,255:99|1/1:76,18,0:12|0/0:0,187,255:99 6|29941267|.|G|C|999|.|DP=159;VDB=0.0003;AF1=0.33;AC1=2;DP4=105,41,11,1;MQ=50;FQ=999;PV4=0.18,1,1,0.49;CSQ=C|5_prime_UTR_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding|1/10||||8|||||rs28749141||1|HGNC|HGNC:4931||||C:0.3168|C:0.30|C:0.33|C:0.28||C:0.35|||||||||||GT:PL:GQ|0/0:0,247,255:99|1/1:208,23,0:17|0/0:0,187,255:99 6|29941293|.|T|C|999|.|DP=182;VDB=0.0019;AF1=0.3333;AC1=2;DP4=118,44,14,5;MQ=51;FQ=999;PV4=1,1,1,0.5;CSQ=C|5_prime_UTR_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding|1/10||||34|||||rs9260079||1|HGNC|HGNC:4931||||||||||||||||||||GT:PL:GQ|0/0:0,255,255:99|1/1:255,57,0:51|0/0:0,211,255:99 6|29941294|.|G|A|999|.|DP=180;VDB=0.0027;AF1=0.3333;AC1=2;DP4=71,16,61,31;MQ=51;FQ=999;PV4=0.027,7.8e-66,0.34,0.01;CSQ=A|5_prime_UTR_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding|1/10||||35|||||rs9260080||1|HGNC|HGNC:4931||||||||||||||||||||GT:PL:GQ|1/1:255,255,0:99|0/0:0,57,255:57|0/0:0,161,255:99 6|29941376|.|G|T|999|.|DP=195;VDB=0.0063;AF1=0.3333;AC1=2;DP4=106,67,14,6;MQ=54;FQ=999;PV4=0.63,0.068,0.46,1;CSQ=T|intron_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||1/9||||||||rs9260081||1|HGNC|HGNC:4931||||G:0.3232|T:0.69|T:0.74|T:0.72||T:0.61|||||||||||GT:PL:GQ|0/0:0,255,255:99|1/1:255,60,0:54|0/0:0,208,255:99 6|29941404|.|A|C|999|.|DP=208;VDB=0.0134;AF1=0.6667;AC1=4;DP4=46,23,75,59;MQ=54;FQ=212;PV4=0.17,1.9e-100,1e-06,0.48;CSQ=C|intron_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||1/9||||||||rs1632889||1|HGNC|HGNC:4931||||A:0.1579|C:0.78|C:0.82|C:0.95||C:0.80|||||||||||GT:PL:GQ|1/1:200,255,0:99|1/1:182,72,0:72|0/0:0,147,255:99 6|29941420|.|G|C|999|.|DP=220;VDB=0.0173;AF1=0.6667;AC1=4;DP4=48,27,73,63;MQ=57;FQ=212;PV4=0.19,3.5e-81,3e-06,1;CSQ=C|intron_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||1/9||||||||rs1655919||1|HGNC|HGNC:4931||||G:0.0955|C:0.94|C:0.89|C:0.98||C:0.83|||||||||||GT:PL:GQ|1/1:255,255,0:99|1/1:243,72,0:72|0/0:0,199,255:99 6|29941428|.|C|T|999|.|DP=208;VDB=0.0181;AF1=0.3333;AC1=2;DP4=63,33,58,50;MQ=57;FQ=999;PV4=0.089,5.1e-68,0.0018,1;CSQ=T|intron_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||1/9||||||||rs1632888||1|HGNC|HGNC:4931||||T:0.1414|T:0.07|T:0.08|T:0.20||T:0.17|||||||||||GT:PL:GQ|1/1:204,255,0:99|0/0:0,87,255:87|0/0:0,179,255:99 6|29941440|.|T|G|84.2|.|DP=204;VDB=0.0177;AF1=0.3333;AC1=2;DP4=57,36,53,53;MQ=57;FQ=86;PV4=0.12,3.8e-94,0.0029,0.45;CSQ=G|intron_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||1/9||||||||rs1632887||1|HGNC|HGNC:4931||||||||||||||||||||GT:PL:GQ|1/1:129,255,0:99|0/0:0,99,255:99|0/0:0,161,255:99 6|29941442|.|T|C|95.2|.|DP=211;VDB=0.0171;AF1=0.3333;AC1=2;DP4=56,37,32,36;MQ=58;FQ=97;PV4=0.11,1.5e-99,0.14,0.11;CSQ=C|intron_variant|MODIFIER|HLA-A|ENSG00000206503|Transcript|ENST00000396634|protein_coding||1/9||||||||rs1632886||1|HGNC|HGNC:4931||||||||||||||||||||GT:PL:GQ|1/1:140,202,0:99|0/0:0,99,255:99|0/0:0,164,255:99
このように、"HLA-A"を含むすべてのSNPsの行が抽出されています。
(4) grepによるデータの抽出その2: 特定の性質を持つSNPs
特定の性質を持つSNPsを、consequencesなどの属性をキーにして抽出することができます(SNPごとの属性についてはこのエントリを参照)。例えば、ナンセンス変異 (stop_gained)になるSNPをすべて抽出する場合、ggrepで以下のように実行します。
ggrep stop_gained MHC_test4.var.annotated.vcf > stop_gained.vcf
あるいは、ミスセンス変異のSNPsの場合、
ggrep missense MHC_test4.var.annotated.vcf > missense.vcf
(5) grepによるデータの抽出その3: 有害な (deleterious) 変異
SIFTやPolyPhenなどにより予測された、塩基置換による個体への影響をキーにして、特定のSNPsを抽出することもできます(SIFTやPolyPhenについては、以下のエントリを参照)。
たとえば、有害 (deleterious)な変異を起こすSNPsを抽出するには、以下のようにします。
ggrep deleterious MHC_test4.var.annotated.vcf > deleterious.vcf
(6) grepによるデータの抽出その4: 複数の遺伝子上にあるSNPsを同時に抽出する
たとえば、特定の疾患に対して複数の関連遺伝子が存在していて、それらの遺伝子上にあるSNPsを同時に抽出したいような場合、以下のような方法が使えます。
まず、最初に関連遺伝子をリストアップしたファイルを作ります。たとえば、
HLA-A HLA-B HLA-DRB5 HLA-DQB1
これを、たとえばファイル名"candidates.txt"として保存します。
そして、以下のように実行します。
ggrep -wFf candidates.txt MHC_test4.var.annotated.vcf > candidates.vcf
4つの遺伝子上に存在するSNPsのリストになっています(確認して下さい)。
さらに、ここから絞り込むこともできます。たとえば、genotypeがref/alt allelesのheterozygoteになっているものに絞る場合は、以下のようにします。
ggrep 0\/1 candidates.vcf > candidates.hetero.vcf
抽出したSNPsのリストは"|"で区切られたファイルになっています。そのままでは見辛いので、Excelに読み込んで開くと良いです。
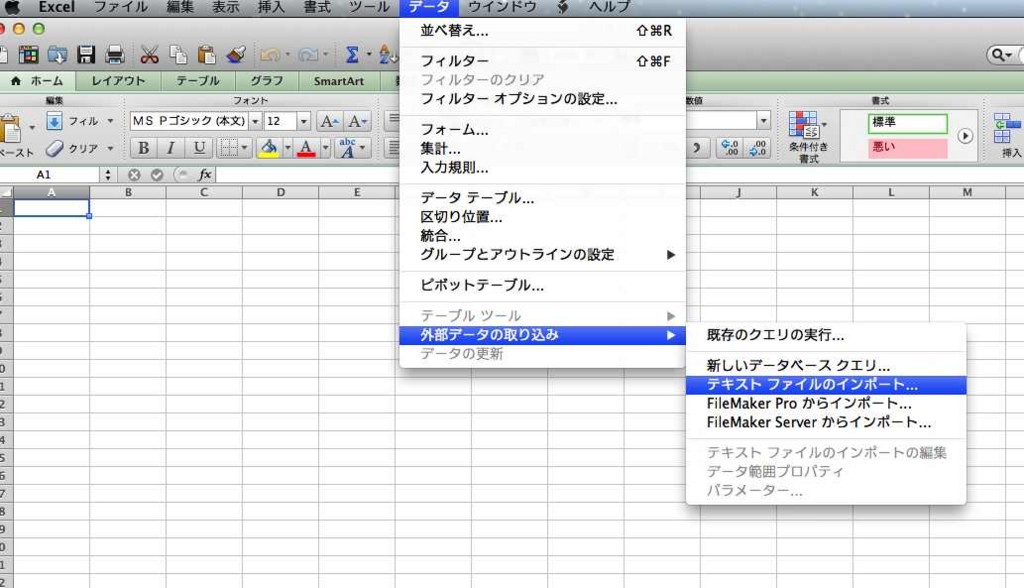
Excelで新規書類を開き、
[データ] --> [外部データの取り込み] --> [テキストファイルのインポート]
を選択します(下図)。
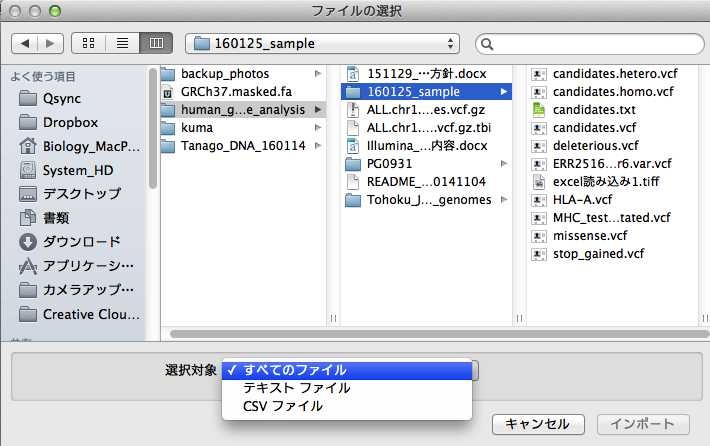
選択対象を「すべてのファイル」にし、読み込むVCFファイルを選択して「インポート」をクリックします(下図)。
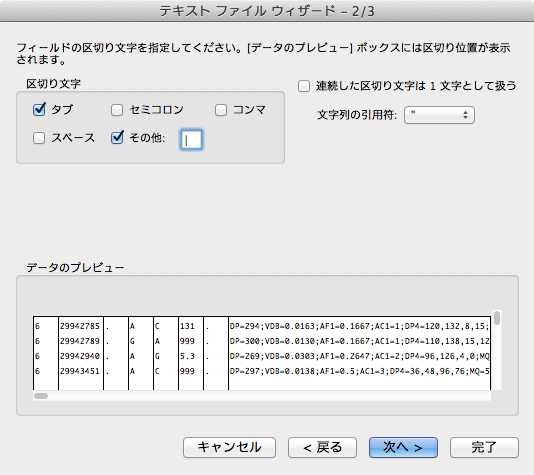
テキストファイルウィザードの最初の画面は、そのまま「次へ」をクリックします。2つ目の画面で、「区切り文字」のところで「その他」をチェックし、区切り文字入力場所に"|"を入力(下図)し、「次へ」をクリックします。
あとはそのまま進めば完了です。