2015-05-18
Variant Effect PredictorによるSNPのアノテーション結果について説明します。
3. アノテーション結果を見る
(1) Summary statistics
解析がうまく行くと、このような結果の画面を見ることができます。
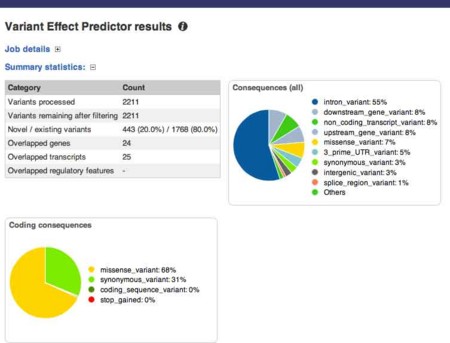
ここでの表と円グラフは、結果全体の要約になっています。"Consequences (all)"は、遺伝子以外の領域も含めたすべてのSNPの位置の存在比率を、"Coding consequences"は遺伝子コード領域のSNPにおける同義・非同義、ナンセンス変異などの存在比率を示しています。
(2) 結果の絞り込み
これら"Summary statistics"の下の部分には、各SNP(多型)ごとのアノテーション結果が、表として示されています。ただし、SNPの数が多い場合には、結果そのままの状態では必要な情報を得ることが難しいです。そこで、表の上にある設定を行う部分(下図)を使用して、必要なSNP情報の絞り込みや、必要なデータのダウンロードを行います。

"Navigation"では、一度に表示するSNPsの数を選択します。Defaultでは5になっているので、もっと多く表示したい場合は、50などに変更します。ただし、SNPsの数が多い(1000以上あるような)場合に"all"を選択してしまうと、表示に時間がかかってしまう場合があります。
次に、結果の表の各列について説明します。
Consequence: 多型variantがどのようなタイプの変異を引き起こすのか、についての情報です。intron_variantはintron内の変異、synonymous_variantは同義変異(アミノ酸を変えないコード領域内の変異:コドンの3rd positionの多くや1st positionの一部の変異)、missense_variantはアミノ酸を変える変異、upstream_gene_variantは遺伝子上流(5' UTRなど)の変異、splice_region_variantはexon-intron境界のsplicing siteに生じた変異、stop_gainedはナンセンス変異になります。
Impact: SNPの表現型に対する影響? LOWは表現型にはあまり関係ない(同義変異など)、MODERATEはある程度関係する(アミノ酸置換など)、MODIFIERは現時点ではわかりません(<--あとで調べること)。
Gene: ENSEMBL gene IDのようです。リンクになっています。
Amino acids, Codons: コード領域のSNPについて、アミノ酸(ミスセンス変異の場合)およびコドンの変異を示しています。
Existing variations: そのSNPが既知の変異に対応する場合、そのSNP IDなどへのリンク。
SIFT: アミノ酸配列の変異が、タンパク質の機能に与える影響を予測するアルゴリズム。予測の方法は、類似した配列を持つ複数のタンパク質の配列の比較に基づいているらしい(詳細はWeb siteを参照)。有害な(deleterious)あるいは許容されない(intolerant)変異は赤で表示されます。
PolyPhen: SIFTと同様に、coding領域の変異がタンパク質の機能に与える影響を予測するツール。SIFTより情報量が多いように見えます。(Website)。
GMAF: 1000 Genomes projectのデータにおけるminor alleleの頻度
AFR-MAF, etc.(その後8列):1000 Genomes projectで調べられている人類集団ごとのSNP allele頻度(existing allele --> majorなほうのallele?)。
これら各列の情報をもとに、表の上部にある設定部分の"Filters"(以下)を使って、必要な情報の絞り込みを行います。
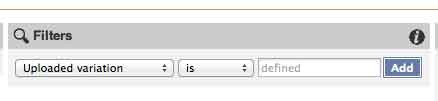
"Consequence"が"missense_variant"である多型のみを表示したい場合、"Uploaded variation"のプルダウンメニューから"Consequence"を選択し、"is" はそのままで、入力部分に"missense_variant"と入力して、"add"をクリックします。
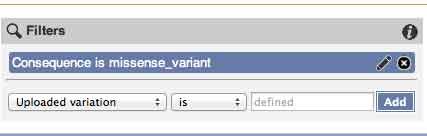
データが絞り込まれ、上のようにFilter(絞り込み)の内容が表示されます。ここから、さらに絞り込むことができます。たとえば、missense_variantのうち、PolyPhemの数値が0.9より大きいものを見たい場合は、"Uploaded variation"のプルダウンメニューから"PolyPhem"を選択し、"is"から">"を選択し、入力欄に0.9を入力して、"add"をクリックします。
Filterの条件を変えるときは、条件の右にある鉛筆を、Filterを削除する場合は、×をクリックします。
アノテーション結果のダウンロード、Rでの編集については、次回に続きます。