次に、IGVで実際にリファレンスゲノムにreadsがマップされた結果を見てみます。
まず、IGVを開きます。デスクトップ上のIGVのエイリアスをダブルクリックします。このとき、IGVでは"Loading genome"という表示が一瞬出ますが、このときリファレンスゲノムとしてhg19を読み込んでいるようです。
IGVが起動したら、自分のサンプルを読み込みます。
(1) BAM fileを読み込む
"File"メニューから"Load from File..."を選択し、開きたいBAM file(indexあり)を選択します。
Mappingされた状態の見方ですが、まず画面左上の2番目の選択する場所(図1)で、"All"から"chr1"などに選択を変更します。
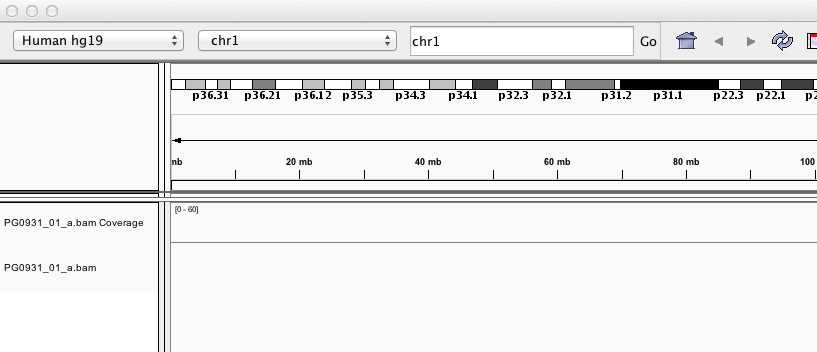
図1. IGVの染色体番号を選択する部分
次に、IGV画面右上のZoomを選択する部分(図2)で、倍率を適当に上げていくと(+側に動かす)、BAMファイルに記録されている個々のreadの位置が表示されます。
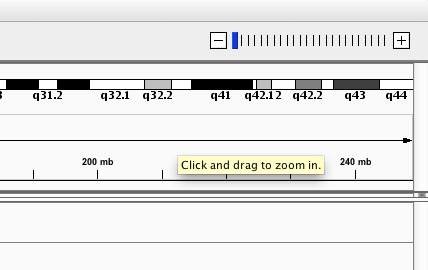
図2. IGVの右上(Zoom)
ただし、最初は見ている染色体上の位置が、何もないところ(セントロメア上)になっているので、赤で示されているカーソルを適当な位置に動かします(図3)。
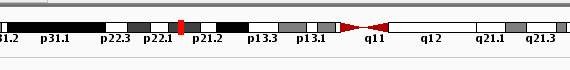
図3. 染色体の位置とカーソル(赤い四角)
(2) VCF file(SNP等のアノテーション)を読み込む
"File"メニューから"Load from File..."を選択し、開きたいVCF fileを選択します。VCFファイルは圧縮された状態(.gz)をそのまま開けます(解凍する必要なし)。
うまく開けると図4のような状態になります。
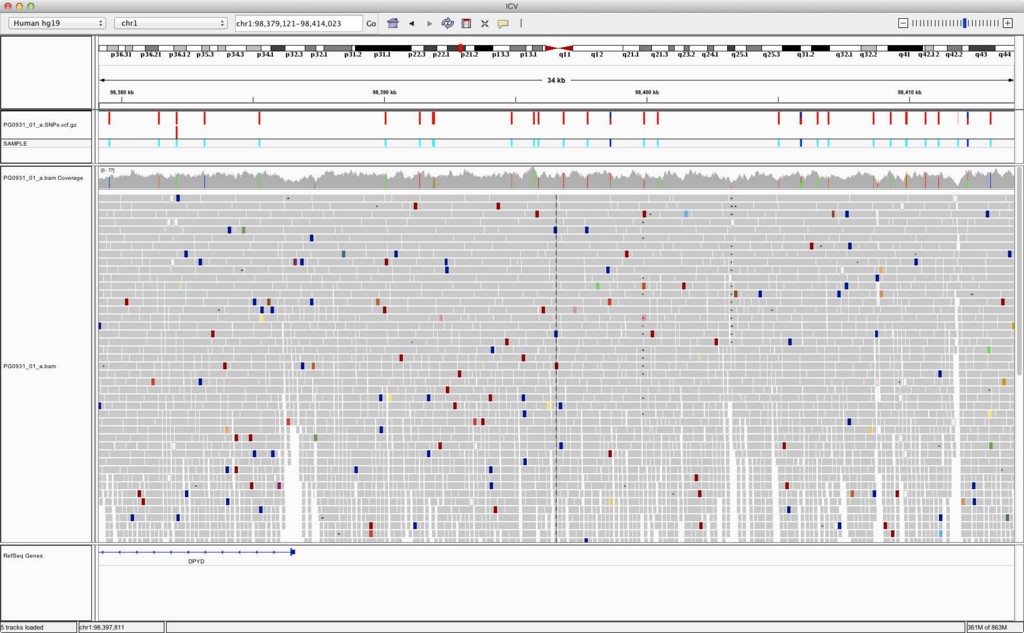
図4. IGV window (BAM fileとVCF fileを読み込んだ状態)
"Sample"の行に示されているのがreferenceのgenotypeの状態で、水色はreferenceと異なるalleleのhomozygote, 青はheterozygoteになっています。色の部分にカーソルを合わせると、各SNPの情報を見ることができます(おそらくVCF fileに記載されているもの)。
IGVの具体的な使い方については次回以降のエントリで説明します。