IGVにRefSeqなど、ヒトゲノム上の遺伝子アノテーションを取り込む方法についてです。
IGVでは、"File" メニューの"Load from Server"から、ゲノム上の位置に関連付けられたアノテーション情報を読み込むことができます。
(1) "File" -> "Load from Server"の中身について
このメニューを選択すると、以下のような画面が出ます。
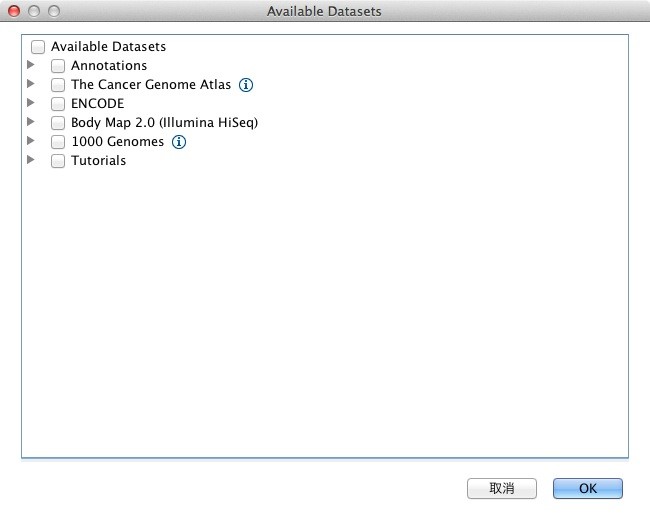
このうち、Available Datasetの一番上にある"Annotations"の中に、いくつかの呼び出し可能なデータがあります。"Annotations"の左側の三角をクリックすると、下図のようにデータの各項目が出てきます。

さらに各項目の左側の三角をクリックすると、各データ内の各項目が出てきます(下図)。たとえば"Genes"の項目の中にも、"Ensembl Genes"や"UCSC Genes"など、異なる由来のアノテーションが含まれます。

(2) "Annotations"の各項目について
- Genes: 遺伝子のアノテーション。EnsemblやUCSCなど、複数の異なる機関によるアノテーションが存在する。通常はどれか一つでOK。
- Phenotype and Disease Associations: 表現型および病気との関連(直訳)、"OMIM Genes"は遺伝子、"GWAS catalog"はGenome wide association studiesの情報で、SNPsです。
- Sequence and Regulation: CpG islandの位置及び領域ごとのGC%を表示します。"GC percentage"がわりと重いです。要注意。
- Variation and Repeats: "dbSNP"はSNPsのデータベースで、"Repeat Masker"はそのまま、RepeatMaskerで予測した繰り返し配列の位置を表示します。
- Comparative Genomics: これについては別のエントリで詳細を見てみます(予定)
(3) 一般的な注意
"File" -> "Load from Server"でデータを取り込む際に、データ項目のtreeの上流の方でチェックを入れてしまうと(たとえば、"Annotations"そのものにチェックを入れる)、膨大なデータを取り込もうとするので、IGV自体が止まってしまいます。ただ、これはちょっとわかりにくい仕様のような気がします。IGVのdocumentsを見ると、ちゃんと以下のような注意書きがあるのですが。
Warning: Selecting a folder selects all of its subfolders and all of the datasets in those folders. This can potentially be a huge amount of data. To be sure you are loading only the data you want, open collapsed folders and select only the datasets of interest.
和訳- 注意:一つのフォルダを選択すると、そのフォルダ内のすべてのサブフォルダ及びそれらのフォルダ内のすべてのデータセットを選択してしまいます。この場合、潜在的に膨大な量のデータになることがあります。すべてのフォルダを開いて、必要なデータセットだけを選択して、必要なデータのみを読み込むよう、気を付けて下さい。