2023年になりました。今年もよろしくお願いします。年末年始は、2年ぶりに広島の実家に帰省して過ごしました。実家では、なんというか息子の成長を感じました。ところで、帰省中の空いた時間に読んでいた、生物の新種発見についての本がとても面白かったです。またブログに感想を書きたいと思います。(2023.1.4)
自分が嫌いだけど好きだった、若い頃の自分へ
斉藤環さんの「自傷的自己愛」の精神分析、とても面白い本だった。この本で書かれている自傷的自己愛というのは、自己評価が低くて、自分はダメな人間だ、とずっと思いながらも、そんな自分自身のことをずっと考えているような心の状態のことで、うつ状態や引きこもりの人によくみられる心の状態のことだ。
著者は専門の精神分析の視点から、自己愛とは何か、また時代と共に変化していく若い人の自己愛の在り方、みたいな話に切り込んでいく。さらに、自傷的自己愛の状態に陥った人がどのように健全な自己愛を取り戻したら良いのか、臨床家らしく具体的な対応手段に話は進んでいく。
この本、全体的に気づきが多く、腑に落ちる部分が多かった。著者は、自己評価が低く、うつ状態にある人が、何か文章を書くなど「何かを作る」ことに関して生産的であることが少なくないことから、自己評価の低さやうつ的な状態は、必ずしも否定されるものではない、と述べる。これはとてもよくわかるところで、確かに気分が落ち込んでいる時って、妙に論文が書けたり、自身の生産性が上がるというか、書くことが一種の治療になっている、という感じがする。
その上で、人間の気分の総和は基本的に一定であり、何らかの自己啓発的な手法で高められた自己肯定感は必ずしも長続きしない、と指摘する。これも理解できるところで、まあ、自己啓発で性急に自己肯定感を高めてもすぐにもとに戻ってしまいそうだし、下手したらカルトや陰謀論にはまってしまいそうだ。
この本の中で、特に個人的に共感したのは、第4章の「優生思想」についての考察の部分だ。自傷的自己愛を持つ人に典型的な考え方として、「仕事での成功」などによって自分の価値を高め、自分を肯定できるようになりたい、というものがある。しかしこの考えは時にとても危険で、仕事上の達成はあまり自己肯定感を高めないばかりか、達成できない時に、「自分には価値がない」と思い込み、そこから「価値のない自分は死んだ方が良い」という「優生思想」に陥ってしまう、という。優生思想とは「生きる価値のない人間が存在する」という考え方のことで、最近は著名な学者とかでも、公に優生思想を語ったりするけれど、自己評価の低い人が、自身を否定することで優生思想に近づいてしまう、というのはとてもありがちなことだ。私自身も、若い時には自己評価が低く、「自分なんて生きる価値がないのではないか」とか思っていたことがある。でもある時、その考えって一種の優生思想だよな、と気づいて、世の中には「自分も含めて」生きる価値がない人間なんていない、と思い直した経験があるのだけど、それを明確に指摘した文章を読んだのは初めてだったので、その点は非常に感心した。
著者も、(多分)言いたいと思っていることは、優生思想を否定することは単純に倫理的な問題ではなくて、あるいは「きれいごと」ではなくて、この世界でなにがあっても、生きている限りは生きていくという「覚悟」なのではないか、ということだ。著者の言葉を引用すると、
「あなたが他者をむやみに否定しない倫理的理由があるというのなら、まったく同じ理由で、自分自身も否定しないでほしい、ということです」(196ページ)
また、この本では、第二章の「コミュニケーション力が何よりも重視される(ハイパー・メリトクラシー)現代」に対する批判、も興味深く読んだ。いつも感じていることだけど、本当に、今の時代に十代を過ごさないといけない若い人たちは大変だよな、とつくづく思う。
若い時の私のような、自己評価が低くて自信を失いがちな若い人たちには、ぜひ読んでほしいと思った。テレビやネットで平然と優生思想を語る、コミュ力に長けた学者や起業家(といわれる人たち)に引っかからないためにも・・・
GPUを使ったNanoporeのベースコール比較
NanoporeのMinIONによるロングリードのゲノム決定において、解析のネックとなるのはベースコールであろう。10-20 Gbpのデータであると、outputのFAST5ファイルからベースコールを行うと、guppyのCPU版だと1ヶ月くらいかかることがある。
しかしながら、GPUをベースコールに使用することで、このステップを非常に高速化することが可能になる。まあ、MinION Mk1Cを使用できれば良いけれど、これは予算的に結構大変である。今回、LinuxのノートパソコンでeGPUを導入することで、ベースコールを高速化することに成功したので紹介する。また、Nanoporeが提供している2種のbasecallerであるguppyとdodadoについて、同一データによる比較を行なったので、その結果を以下にまとめた。
解析環境
下準備
必要な環境
- Python 3.9
- SAMtools version 1.10
- BEDtools
guppy, doradoのインストール
Nanopore communityからプログラムをダウンロード、インストールする
eGPUの設定
以下のエントリを参照
テストデータ
MinIONで取得した某魚類のゲノムデータのFAST5ファイル:約2 Gbp(約20万リード) 1,000 bp未満の配列は、ナノポアシークエンスの時点で捨てている
ベースコール
1. Guppy (Version6.4.6+ae70e8f)
FAST5ファイルに対して、以下のコマンドを実行する。
# -x オプションはeGPUの番号("nvidia-smi"で確認)を指定 guppy_basecaller --flowcell FLO-MIN106 --kit SQK-LSK109 -x cuda:0 -i fast5/ -s fastq -r
2. dorado (Version 0.1.1)
doradoでは、FAST5形式の代わりに、Nanoporeの新しいデータフォーマットであるPOD5形式を使用する。そこで最初に、このサイトに従ってPOD5のconverterをpip installし、ファイルの変換を行う。
# install pod5 converter pip install pod5 # pod5 convert pod5 convert fast5 ./fast5/*.fast5 pod5 --output-one-to-one fast5/
doradoではベースコールのモデルをMinIONのプロトコルに応じて選択する必要があるので、適切なモデルをダウンロードする。たとえば今回の"--flowcell FLO-MIN106 --kit SQK-LSK109"のケースであれば、"dna_r9.4.1_e8_hac@v3.3"を使用する。
# dorado download dorado download --model dna_r9.4.1_e8_hac@v3.3
POD5 fileに対して、doradoを実行する。
# dorado basecaller # デフォルトではsamファイルがoutputとして出力される。ファイルサイズが大きくなるので、samtoolsを使ってBAMに変換する dorado basecaller dna_r9.4.1_e8_hac@v3.3 pod5/ | samtools view -Sb -@ 4 > 230119_1.bam
Outputのunaligned BAMファイルから、BEDtoolsでFASTQ fileを書き出す。
# bedtools bedtools bamtofastq -i 230119_1.bam -fq 230119_1.dorado.fastq
ベンチマーク比較
計算時間とデータ出力
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| Run time | 1h50m | 1h05m |
| Mean read length | 13,480.70 | 13,180.60 |
| Mean read quality | 13.9 | 12.6 |
| Median read length | 6,988.00 | 6,789.00 |
| Median read quality | 14.2 | 13.4 |
| Number of reads | 164,352.00 | 199,115.00 |
| Read length N50 | 26,971.00 | 26,390.00 |
| STDEV read length | 15,284.40 | 15,292.00 |
| Total bases | 2,215,586,980.00 | 2,624,461,937.00 |
Number, percentage and megabases of reads above quality cutoffs
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| >Q5 | 164352 (100.0%) 2215.6Mb | 196617 (98.7%) 2600.7Mb |
| >Q7 | 164352 (100.0%) 2215.6Mb | 181224 (91.0%) 2433.3Mb |
| >Q10 | 155864 (94.8%) 2098.3Mb | 154916 (77.8%) 2080.9Mb |
| >Q12 | 128819 (78.4%) 1746.4Mb | 127150 (63.9%) 1720.1Mb |
| >Q15 | 58331 (35.5%) 788.5Mb | 54200 (27.2%) 715.9Mb |
Top 5 highest mean basecall quality scores and their read lengths
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| 1 | 25.5 (1143) | 27.8 (1568) |
| 2 | 22.0 (1305) | 24.4 (1791) |
| 3 | 21.7 (1360) | 23.5 (1033) |
| 4 | 21.6 (916) | 23.2 (1344) |
| 5 | 21.3 (1286) | 22.1 (1303) |
Top 5 longest reads and their mean basecall quality score
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| 1 | 202700 (10.3) | 1232223 (4.5) |
| 2 | 183757 (12.1) | 202799 (10.1) |
| 3 | 168279 (9.8) | 184101 (11.8) |
| 4 | 158922 (10.8) | 168920 (9.4) |
| 5 | 156477 (11.1) | 159142 (10.5) |
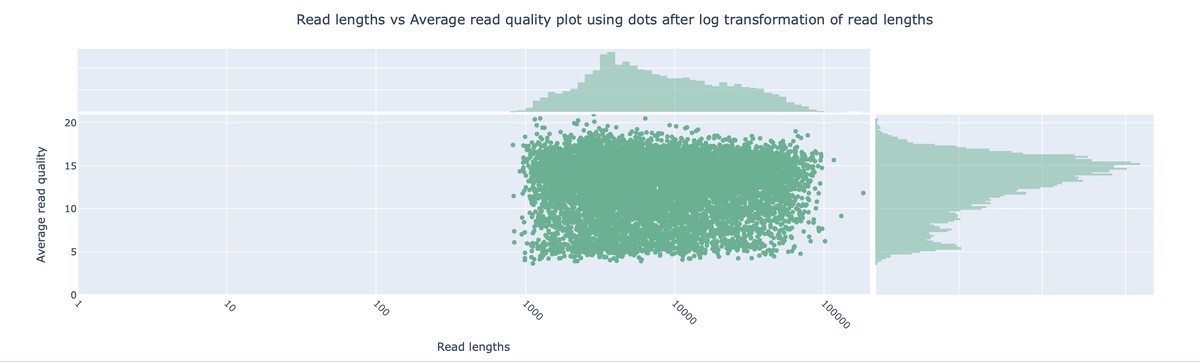
リード長とリードクオリティのプロット
1. Guppy

2. dorado
ベンチマーク結果まとめ
計算時間はdoradoがguppyよりやや速かった(約1.7倍)。データ量もdorado > Guppyであるが、これはGuppyが、デフォルト設定で平均Q < 10のリードをFail判定するためと思われる。プロットでもGuppyではQ < 10のリードはほぼ存在しない。doradoではこれらのリードも含むために、ややデータ量は多く、また平均リード長やquality, N50などが若干低くなると考えられる。
また、doradoの方では1つだけかなり長いリード(>1 Mb)が含まれていた。このリードは平均Q-valueが低いので、guppyでは除かれてしまうようだ。
Ubuntu 20.04を入れたノートパソコンでeGPUを使えるようにする
Ubuntu (20.04 LTS)を入れたノートパソコンでeGPUのセッティングをしたので、備忘録的なメモを書いておく。
eGPUの主な使用目的
- Nanopore MinIONで読んだゲノム配列のベースコール(guppy_basecaller, dorado)
- NextGenMapなどによるショートリードのリファレンスゲノムへのマッピング
使用機材
- 使用マシン:マウスコンピュータ DAIV 3n(WIndowsは削除し、Ubuntu 20.04 LTSをクリーンインストールしたもの) Thunderbolt 3 対応のノートパソコンなら何でも
- eGPU box: AKTIO Node TITAN
- GPU: NVIDIA Quadro RTX 4000
事前準備
anacondaを使用できるようにしておく。
作業手順
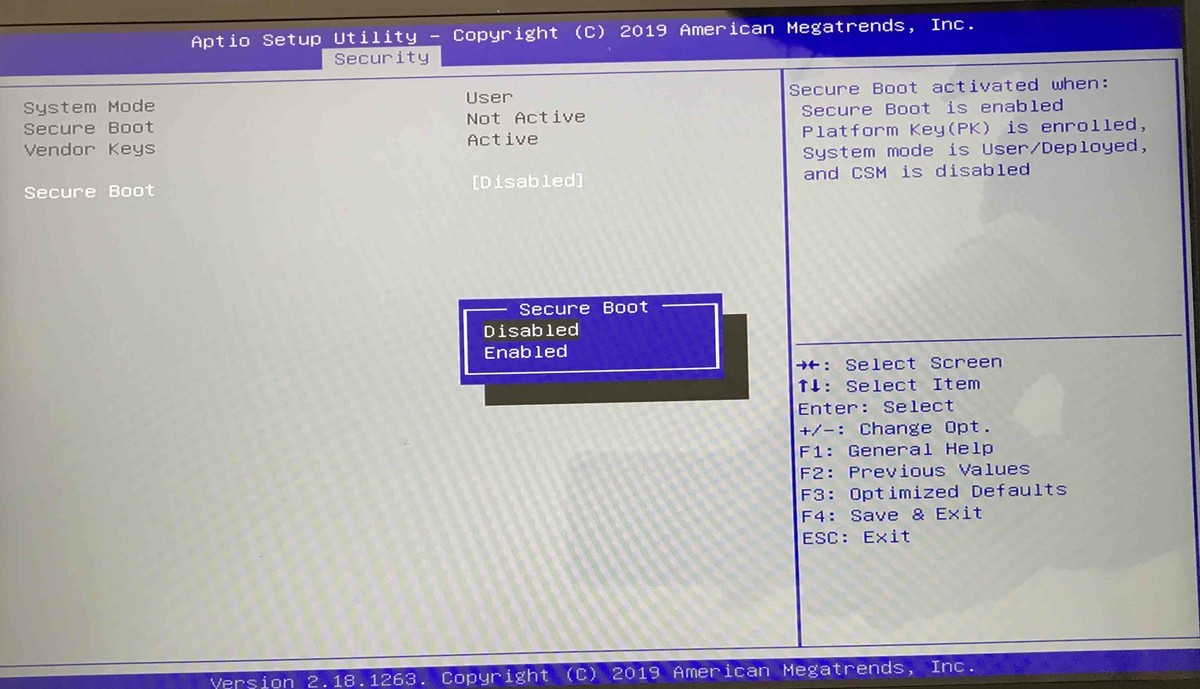
1. Secure bootのオフ
Ubuntuのターミナルに以下のように入力してBIOSを起動し、Secure bootを"Disabled"に変更する。
sudo systemctl reboot --firmware-setup
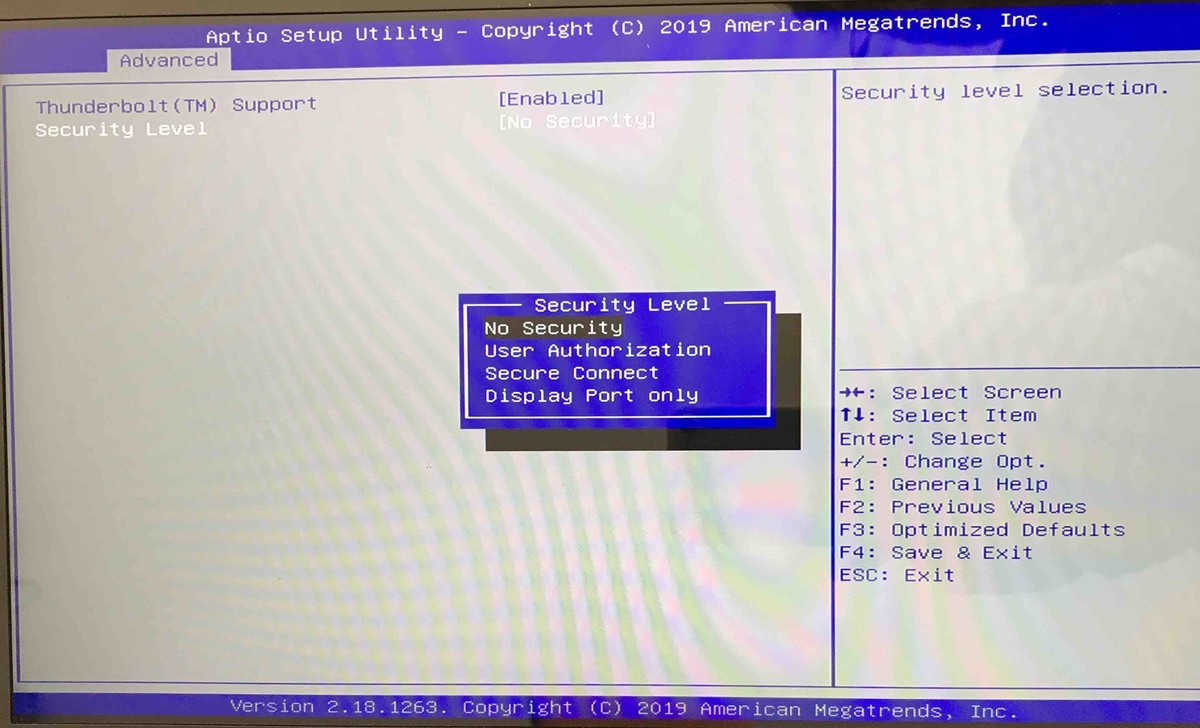
2. Thunderbolt 3のセキュリティをオフ(こちらは不要かもしれない:未確認)
次にBIOSからThunderbolt 3のセキュリティをオフにする。設定を変更して再起動(Save & Exit)。
3. eGPU Boxを接続する
Thunderbolt 3のケーブルをパソコンに繋いで、eGPU boxの電源を入れる。
4. NVIDIAのデバイスドライバをインストールする
当然、ドライバが入っていない状態で接続してもコンピュータはeGPUを認識しないので、まずデバイスドライバをインストールする。以下のコマンドを実行する。
sudo apt install nvidia-driver-440
インストールが終了したら、コンピュータを再起動する。
5. CUDA-Toolkitをインストールする
いろいろな方法があるが、今回はcondaを使って導入した(これが一番簡単だと思う)。以下を実行する。現在はCUDA 11.3.1をインストールした。使用するGPUによって適切なバージョンを選択する。
conda install -c anaconda cudatoolkit
2022-12-26の近況
とても忙しかった2022年も、残すところあと1週間になりました。最近、自分の研究に対するモチベーションが上がっているな、と感じます。あとは頑張って研究を進めて、論文を書くのみであります(2022.12.26)
