NanoporeのMinIONによるロングリードのゲノム決定において、解析のネックとなるのはベースコールであろう。10-20 Gbpのデータであると、outputのFAST5ファイルからベースコールを行うと、guppyのCPU版だと1ヶ月くらいかかることがある。
しかしながら、GPUをベースコールに使用することで、このステップを非常に高速化することが可能になる。まあ、MinION Mk1Cを使用できれば良いけれど、これは予算的に結構大変である。今回、LinuxのノートパソコンでeGPUを導入することで、ベースコールを高速化することに成功したので紹介する。また、Nanoporeが提供している2種のbasecallerであるguppyとdodadoについて、同一データによる比較を行なったので、その結果を以下にまとめた。
解析環境
下準備
必要な環境
- Python 3.9
- SAMtools version 1.10
- BEDtools
guppy, doradoのインストール
Nanopore communityからプログラムをダウンロード、インストールする
eGPUの設定
以下のエントリを参照
テストデータ
MinIONで取得した某魚類のゲノムデータのFAST5ファイル:約2 Gbp(約20万リード) 1,000 bp未満の配列は、ナノポアシークエンスの時点で捨てている
ベースコール
1. Guppy (Version6.4.6+ae70e8f)
FAST5ファイルに対して、以下のコマンドを実行する。
# -x オプションはeGPUの番号("nvidia-smi"で確認)を指定 guppy_basecaller --flowcell FLO-MIN106 --kit SQK-LSK109 -x cuda:0 -i fast5/ -s fastq -r
2. dorado (Version 0.1.1)
doradoでは、FAST5形式の代わりに、Nanoporeの新しいデータフォーマットであるPOD5形式を使用する。そこで最初に、このサイトに従ってPOD5のconverterをpip installし、ファイルの変換を行う。
# install pod5 converter pip install pod5 # pod5 convert pod5 convert fast5 ./fast5/*.fast5 pod5 --output-one-to-one fast5/
doradoではベースコールのモデルをMinIONのプロトコルに応じて選択する必要があるので、適切なモデルをダウンロードする。たとえば今回の"--flowcell FLO-MIN106 --kit SQK-LSK109"のケースであれば、"dna_r9.4.1_e8_hac@v3.3"を使用する。
# dorado download dorado download --model dna_r9.4.1_e8_hac@v3.3
POD5 fileに対して、doradoを実行する。
# dorado basecaller # デフォルトではsamファイルがoutputとして出力される。ファイルサイズが大きくなるので、samtoolsを使ってBAMに変換する dorado basecaller dna_r9.4.1_e8_hac@v3.3 pod5/ | samtools view -Sb -@ 4 > 230119_1.bam
Outputのunaligned BAMファイルから、BEDtoolsでFASTQ fileを書き出す。
# bedtools bedtools bamtofastq -i 230119_1.bam -fq 230119_1.dorado.fastq
ベンチマーク比較
計算時間とデータ出力
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| Run time | 1h50m | 1h05m |
| Mean read length | 13,480.70 | 13,180.60 |
| Mean read quality | 13.9 | 12.6 |
| Median read length | 6,988.00 | 6,789.00 |
| Median read quality | 14.2 | 13.4 |
| Number of reads | 164,352.00 | 199,115.00 |
| Read length N50 | 26,971.00 | 26,390.00 |
| STDEV read length | 15,284.40 | 15,292.00 |
| Total bases | 2,215,586,980.00 | 2,624,461,937.00 |
Number, percentage and megabases of reads above quality cutoffs
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| >Q5 | 164352 (100.0%) 2215.6Mb | 196617 (98.7%) 2600.7Mb |
| >Q7 | 164352 (100.0%) 2215.6Mb | 181224 (91.0%) 2433.3Mb |
| >Q10 | 155864 (94.8%) 2098.3Mb | 154916 (77.8%) 2080.9Mb |
| >Q12 | 128819 (78.4%) 1746.4Mb | 127150 (63.9%) 1720.1Mb |
| >Q15 | 58331 (35.5%) 788.5Mb | 54200 (27.2%) 715.9Mb |
Top 5 highest mean basecall quality scores and their read lengths
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| 1 | 25.5 (1143) | 27.8 (1568) |
| 2 | 22.0 (1305) | 24.4 (1791) |
| 3 | 21.7 (1360) | 23.5 (1033) |
| 4 | 21.6 (916) | 23.2 (1344) |
| 5 | 21.3 (1286) | 22.1 (1303) |
Top 5 longest reads and their mean basecall quality score
| Guppy (Version6.4.6+ae70e8f) | dorado (Version 0.1.1) | |
|---|---|---|
| 1 | 202700 (10.3) | 1232223 (4.5) |
| 2 | 183757 (12.1) | 202799 (10.1) |
| 3 | 168279 (9.8) | 184101 (11.8) |
| 4 | 158922 (10.8) | 168920 (9.4) |
| 5 | 156477 (11.1) | 159142 (10.5) |
リード長とリードクオリティのプロット
1. Guppy

2. dorado
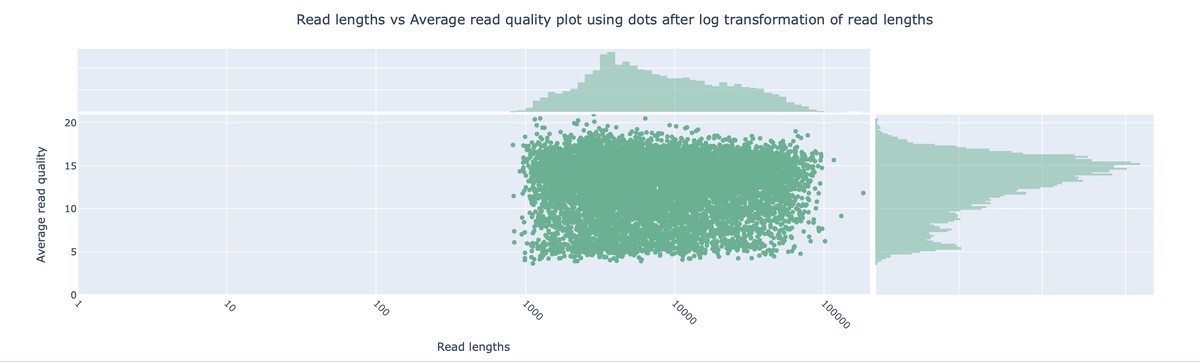
ベンチマーク結果まとめ
計算時間はdoradoがguppyよりやや速かった(約1.7倍)。データ量もdorado > Guppyであるが、これはGuppyが、デフォルト設定で平均Q < 10のリードをFail判定するためと思われる。プロットでもGuppyではQ < 10のリードはほぼ存在しない。doradoではこれらのリードも含むために、ややデータ量は多く、また平均リード長やquality, N50などが若干低くなると考えられる。
また、doradoの方では1つだけかなり長いリード(>1 Mb)が含まれていた。このリードは平均Q-valueが低いので、guppyでは除かれてしまうようだ。